シリーズ ウィズコロナ時代のバイオインフォマティクス
-第1回 単粒子クライオ電子顕微鏡-
文責:がんゲノム医療センター 池田 健
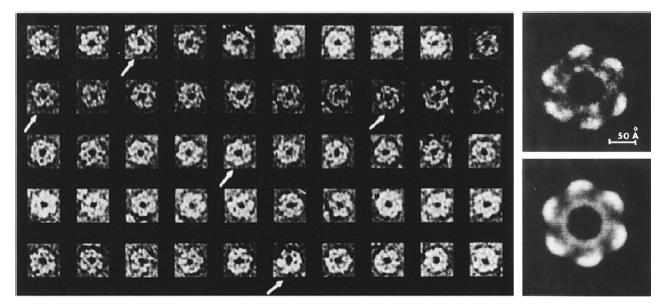
図1:一粒子平均法(Frank, 2018)
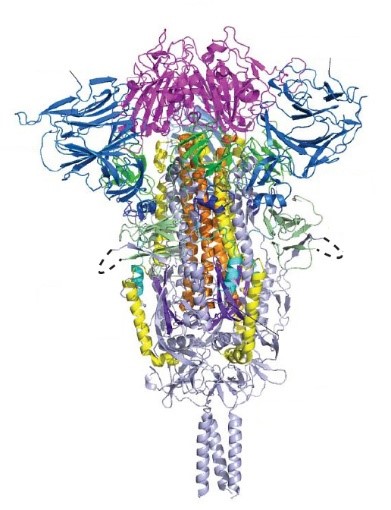
図2:SARS-Cov-2のスパイクタンパク質(Cai, 2020)
タンパク質の立体構造を決定する方法にはX線結晶解析法、核磁気共鳴法および電子顕微鏡法がある。
タンパク質構造データバンク(PDB)に登録される構造は、X線結晶解析法によるものが圧倒的に多い。
しかし、X線結晶解析法では文字通り結晶が必要であり、結晶化できない大きなタンパク質複合体の構造決定には向いていない。
電子顕微鏡の弱点は他の方法に比べ分解能が低い点にあったが、近年、単粒子クライオ電子顕微鏡法によって非常に高い分解能が実現されている(2017年、ノーベル化学賞)。
ブレイクスルーとなったのは、タンパク質粒子の載ったグリッドを液体エタンに投入して瞬間的に凍結させ、粒子をガラス状の薄膜に閉じ込めたことである。
これによってタンパク質の構造を保持しつつ電子線から試料を保護できるようになった。
もうひとつは、コンピュータによる3D構造の再構築である。サンプル中の試料は、グリッド上で同じ分子がランダムな方向に並んだものとみなせる。
同一分子の、多方向からの多数の「スナップショット」を集め、コンピュータ上で平均化し再構築する(図1)。
これらの技術により原子分解能に近い精度が得られるようになった(図2)。
◆参考
Cai、 Y. et al. Distinct conformational states of SARS-CoV-2 spike protein. Science 369: 1586-1592、 2020







